R: Brunner-Munzel検定をためす [統計]
先日の生態学会の粕谷さんの講演でも ふれられていましたが、不等分散のときにMann-WhitneyのU検定(Wilcoxonの順位和検定)をしている例はわりとよく目にします(不等分散のときにつかえない理由はKuboLog 2005-01-29など)。
そういうわけで、Brunner-Munzel検定をためしてみました。やっていることはhoxo_mさんの「マイナーだけど最強の統計的検定 Brunner-Munzel 検定」の後追いです。purrrの練習かもしれません。
まずは正規分布の場合です。平均が同じで分散の異なる分布で、サンプルサイズをかえて ためしてみました。以下はRによるコードです。
library(ggplot2)
library(purrr)
library(dplyr)
library(lawstat)
# 正規分布
test_norm <- function(R = 2000, N = c(30, 30),
mu = c(0, 0), sigma = c(1, 1)) {
y <- purrr::map(seq_len(R), function(i) {
x1 <- rnorm(N[1], mean = mu[1], sd = sigma[1])
x2 <- rnorm(N[2], mean = mu[2], sd = sigma[2])
wl <- wilcox.test(x1, x2)$p.value
bm <- brunner.munzel.test(x1, x2)$p.value
c(wl, bm)
})
df <- data.frame(p_value = purrr::simplify(y),
method = rep(c("Wilcoxon", "BM"), R))
plot <- ggplot(df, aes(p_value, fill = method)) +
geom_histogram(binwidth = 0.05, boundary = -0.5,
position = "dodge")
print(plot)
summary <- df %>%
dplyr::mutate(lt5 = if_else(p_value < 0.05, 1 / (n() / 2), 0)) %>%
dplyr::group_by(method) %>%
dplyr::summarize_at("lt5", sum)
print(summary)
}
set.seed(20180316)
test_norm(R = 5000, N = c(15, 45), mu = c(0, 0), sigma = c(1, 4))
test_norm(R = 5000, N = c(30, 30), mu = c(0, 0), sigma = c(1, 4))
test_norm(R = 5000, N = c(45, 15), mu = c(0, 0), sigma = c(1, 4))
一方の分散を1、他方を4として、p値の分布をシミュレーションで計算しました。
結果です。N = c(15, 45)のとき
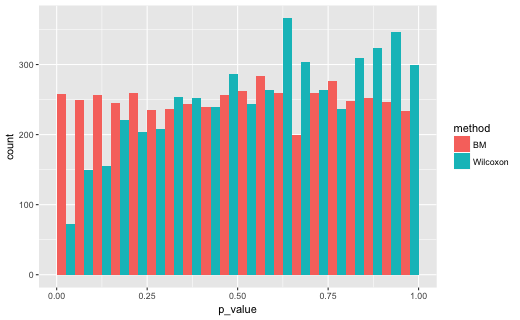
p<0.05となった割合です。
# A tibble: 2 x 2
method lt5
1 BM 0.0516
2 Wilcoxon 0.0144
N = c(30, 30)のとき
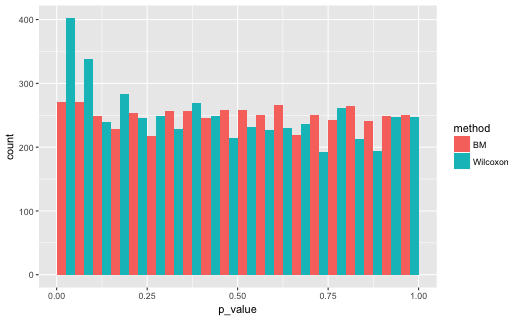
# A tibble: 2 x 2
method lt5
1 BM 0.0542
2 Wilcoxon 0.0804
N = c(45,15)のとき
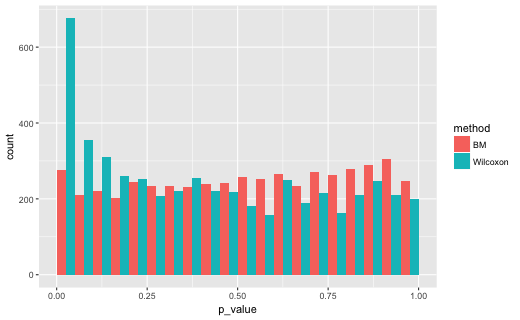
# A tibble: 2 x 2
method lt5
1 BM 0.0552
2 Wilcoxon 0.1352
Brunner-Munzelの方は、p<0.05となる割合がだいたい0.05となっています。一方、U検定(Wilcoxonの順位和検定)の方は、分散とサンプルサイズの組み合わせより変化しました。
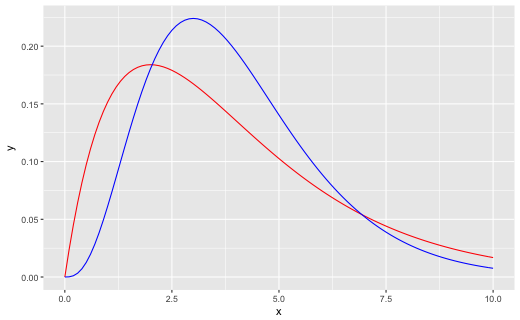
つづいて、ガンマ分布でためしました。下のような分布で比較しました。
# ガンマ分布
shape <- c(2, 4)
scale <- c(2, 1)
ggplot(data.frame(x = seq(0, 10, 0.01)), aes(x)) +
stat_function(col = "red", fun = dgamma,
args = list(shape = shape[1], scale = scale[1])) +
stat_function(col = "blue", fun = dgamma,
args = list(shape = shape[2], scale = scale[2]))
テストコードです。
test_gamma <- function(R = 2000, N = c(30, 30),
shape = c(2, 4), scale = c(2, 1)) {
y <- purrr::map(seq_len(R), function(i) {
x1 <- rgamma(N[1], shape = shape[1], scale = scale[1])
x2 <- rgamma(N[2], shape = shape[2], scale = scale[2])
wl <- wilcox.test(x1, x2)$p.value
bm <- brunner.munzel.test(x1, x2)$p.value
c(wl, bm)
})
df <- data.frame(p_value = purrr::simplify(y),
method = rep(c("Wilcoxon", "BM"), R))
plot <- ggplot(df, aes(p_value, fill = method)) +
geom_histogram(binwidth = 0.05, boundary = -0.5,
position = "dodge")
print(plot)
summary <- df %>%
dplyr::mutate(lt5 = if_else(p_value < 0.05, 1 / (n() / 2), 0)) %>%
dplyr::group_by(method) %>%
dplyr::summarize_at("lt5", sum)
print(summary)
}
set.seed(20180316)
test_gamma(R = 5000, N = c(45, 15), shape = shape, scale = scale)
test_gamma(R = 5000, N = c(30, 30), shape = shape, scale = scale)
test_gamma(R = 5000, N = c(15, 45), shape = shape, scale = scale)
結果です。N = c(45, 15)のとき
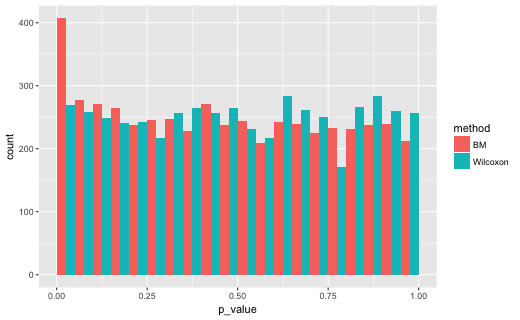
# A tibble: 2 x 2
method lt5
1 BM 0.0814
2 Wilcoxon 0.0538
N = c(30, 30)のとき
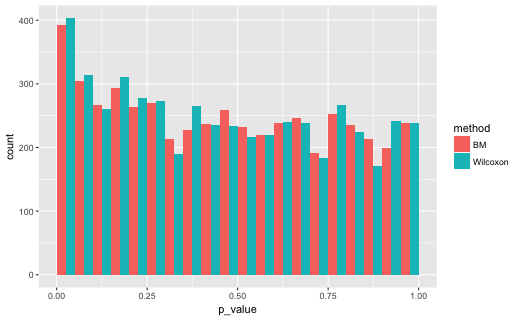
# A tibble: 2 x 2
method lt5
1 BM 0.0786
2 Wilcoxon 0.0806
N = c(15,45)のとき
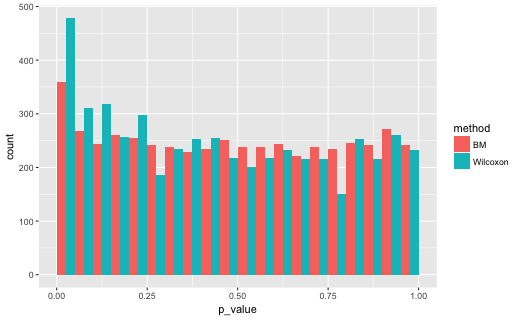
# A tibble: 2 x 2
method lt5
1 BM 0.0720
2 Wilcoxon 0.0956
この場合、比較している分布の中央値は多少ちがっていますし、群間の平均順位も微妙にちがっているかもしれません。Brunner-Munzel検定でp<0.05となった割合が0.07〜0.08となったのはそのためかとおもわれます。一方、U検定(Wilcoxonの順位和検定)では、やはりサンプルサイズと分散の組み合わせにより、p<0.05となった割合が変化しています。
参考にしたところ
- hoxo_m マイナーだけど最強の統計的検定 Brunner-Munzel 検定
- 名取真人 (2014) マン・ホイットニーのU検定と不等分散時における代表値の検定法, 霊長類研究30:173–185
- 奥村晴彦 Brunner-Munzel検定
[追記] Gistにソースをおいて、はりつけてみるテストです。





コメント 0