シカ−ネズミ−ササ−実生系を階層ベイズで解析 [統計]
Itô and Hino (2004)やItô and Hino (2005)の話をパス解析で解析してみようとしていろいろと苦労していたのが去年のいまごろ。結局、多変量正規分布と線形性のしばりがきつくてうまくいかなかった。それを階層ベイズで解析してみようと思った。
まずは、Clark et al. (2004)風の絵を描いてみた。
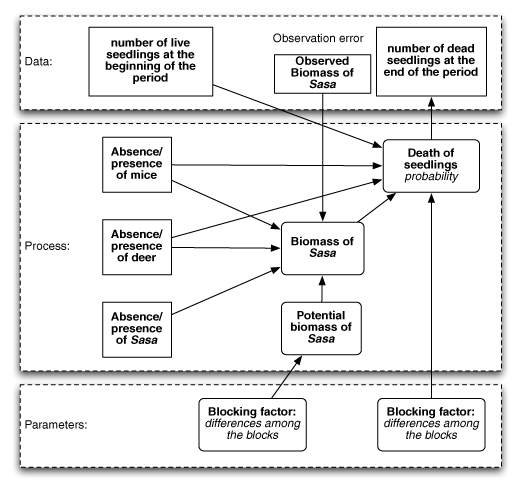
ブロック因子はHyperparameterに置くのが正しい。Parameterには生存率とササ現存量の切片を置く。(3/22追記)
つづいて、コードを書いてみる。
var
n0[N,M], # number of live seedlings at the beginning of the
# period
dead[N,M], # number of dead seedlings at the end of the period
p[N,M], # mortality
deer[N], # absence/presence of deer (0/1)
mice[N], # absence/presence of mice (0/1)
sasa[N], # absence/presence of Sasa (0/1)
dw[N,M], # observed aboveground biomass of Sasa
rdw[N,M], # real aboveground biomass of Sasa
pw, # potential aboveground biomass of Sasa
epsilon.p[M],
epsilon.dw[M],
tau.p, tau.dw, mu,
sigma.p, sigma.dw
model {
for (j in 1:M) {
epsilon.p[j] ~ dnorm(0.0, tau.p);
epsilon.dw[j] ~ dnorm(0.0, tau.dw);
for (i in 1:N) {
dead[i,j] ~ dbin(p[i,j], n0[i,j]);
logit(p[i,j]) <- beta + beta.w * rdw[i,j] +
beta.d * deer[i] + beta.m * mice[i] +
epsilon.p[j];
dw[i,j] ~ dgamma(rdw[i,j] * mu, mu);
rdw[i,j] <- sasa[i] * pw *
exp(beta.ds * deer[i] + beta.ms * mice[i] +
epsilon.dw[j]) + (1 - sasa[i]) * 0.001;
}
}
# priors
beta ~ dnorm(0.0, 1.0E-6);
beta.w ~ dnorm(0.0, 1.0E-6);
beta.d ~ dnorm(0.0, 1.0E-6);
beta.m ~ dnorm(0.0, 1.0E-6);
beta.ds ~ dnorm(0.0, 1.0E-6);
beta.ms ~ dnorm(0.0, 1.0E-6);
pw ~ dgamma(1.0E-6, 1.0E-6);
tau.p ~ dgamma(1.0E-6, 1.0E-6);
tau.dw ~ dgamma(1.0E-6, 1.0E-6);
mu ~ dgamma(1.0E-6, 1.0E-6);
sigma.p <- 1/sqrt(tau.p);
sigma.dw <- 1/sqrt(tau.dw);
}
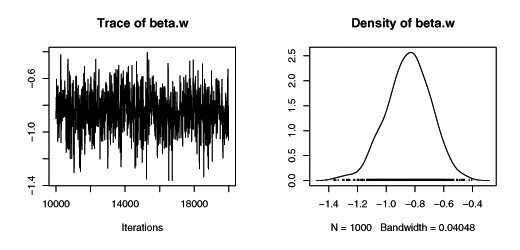
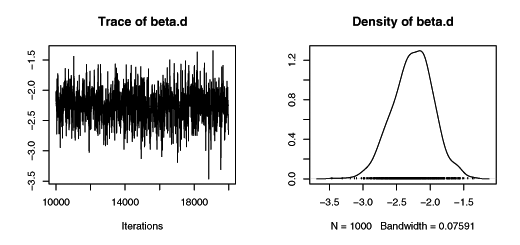
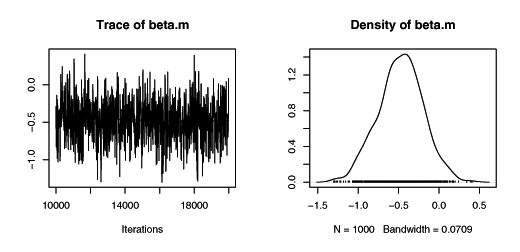
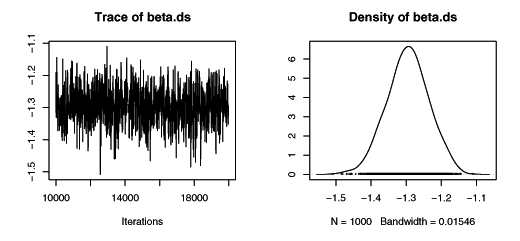
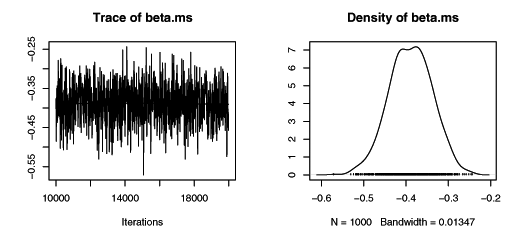
JAGSでMCMC計算をおこなった結果。なんだか妥当な値が出ている。





コメント 0