Stan: はずれ値をあつかう [統計]
『ベイズモデリングの世界』講義3の はずれ値のモデルをStanでやってみました。
Rコードです。データの生成と、はずれ値がない場合とある場合の回帰、グラフの描画までをおこないます。
library(ggmcmc) library(rstan) options(mc.cores = parallel::detectCores()) rstan_options(auto_write = TRUE) set.seed(20180308) N <- 36 X <- runif(N, 0, 20) Y <- X + rnorm(N, 0, 1) ## はずれ値 N2 <- 4 X2 <- c(X, runif(N2, 0, 10)) Y2 <- c(Y, rnorm(N2, 40, 2)) summary(lm(Y ~ X)) summary(lm(Y2 ~ X2)) data.frame(X = X2, Y = Y2) %>% ggplot(mapping = aes(X, Y)) + geom_point()
このようなデータになります。
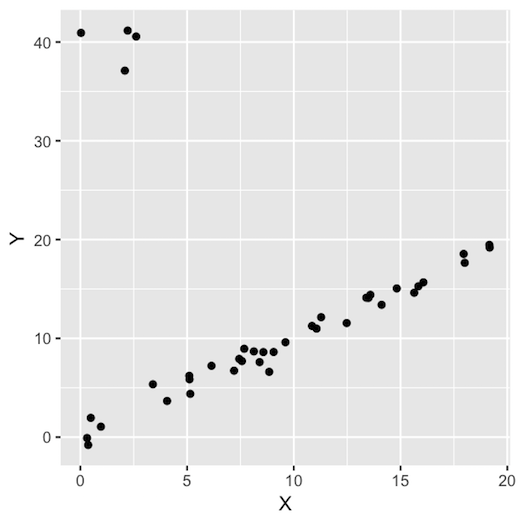
はずれ値のない場合と、ある場合の回帰の結果です。
> summary(lm(Y ~ X))
Call:
lm(formula = Y ~ X)
Residuals:
Min 1Q Median 3Q Max
-2.32861 -0.59270 -0.02385 0.61868 1.74987
Coefficients:
Estimate Std. Error t value Pr(>|t|)
(Intercept) 0.26896 0.29384 0.915 0.366
X 0.98001 0.02641 37.103 <2e-16 ***
---
Signif. codes: 0 ‘***’ 0.001 ‘**’ 0.01 ‘*’ 0.05 ‘.’ 0.1 ‘ ’ 1
Residual standard error: 0.8527 on 34 degrees of freedom
Multiple R-squared: 0.9759, Adjusted R-squared: 0.9752
F-statistic: 1377 on 1 and 34 DF, p-value: < 2.2e-16
> summary(lm(Y2 ~ X2))
Call:
lm(formula = Y2 ~ X2)
Residuals:
Min 1Q Median 3Q Max
-12.599 -5.958 -2.696 1.553 29.166
Coefficients:
Estimate Std. Error t value Pr(>|t|)
(Intercept) 11.7585 3.1425 3.742 0.000603 ***
X2 0.1192 0.2972 0.401 0.690652
---
Signif. codes: 0 ‘***’ 0.001 ‘**’ 0.01 ‘*’ 0.05 ‘.’ 0.1 ‘ ’ 1
Residual standard error: 10.62 on 38 degrees of freedom
Multiple R-squared: 0.004214, Adjusted R-squared: -0.02199
F-statistic: 0.1608 on 1 and 38 DF, p-value: 0.6907
Stanのコードです。このモデルではpを はずれ値である確率としました。はずれ値は、幅のひろい一様分布から生成されたとしています。Stanでは離散パラメーターが つかえないので、周辺化してあります。
data {
int<lower = 0> N;
vector[N] X;
vector[N] Y;
}
parameters {
real alpha;
real beta;
real<lower = 0> sigma;
real<lower = 0, upper = 1> p;
}
model {
for (n in 1:N) {
real lp[2];
lp[1] = bernoulli_lpmf(0 | p)
+ normal_lpdf(Y[n] | alpha + beta * X[n], sigma);
lp[2] = bernoulli_lpmf(1 | p)
+ log(1.0e-4);
target += log_sum_exp(lp);
}
alpha ~ normal(0, 10);
beta ~ normal(0, 10);
sigma ~ normal(0, 10);
}
RからStanを実行します。
stan_data <- list(N = length(X2),
X = X2,
Y = Y2)
fit <- stan("outlier.stan",
data = stan_data,
iter = 2000, warmup = 1000, thin = 1)
print(fit)
結果です。
Inference for Stan model: outlier.
4 chains, each with iter=2000; warmup=1000; thin=1;
post-warmup draws per chain=1000, total post-warmup draws=4000.
mean se_mean sd 2.5% 25% 50% 75% 97.5% n_eff Rhat
alpha 0.27 0.01 0.30 -0.33 0.07 0.27 0.47 0.85 2276 1
beta 0.98 0.00 0.03 0.93 0.96 0.98 1.00 1.03 2354 1
sigma 0.88 0.00 0.11 0.70 0.81 0.87 0.95 1.12 2603 1
p 0.12 0.00 0.05 0.04 0.09 0.12 0.15 0.23 2725 1
lp__ -98.66 0.03 1.37 -102.04 -99.34 -98.37 -97.62 -96.94 1699 1
alpha, beta, sigmaの事後平均は、はずれ値をのぞいた回帰の値とちかくなっています。
つづいて、pを、各データについて推定してみました。
data {
int<lower = 0> N;
vector[N] X;
vector[N] Y;
}
parameters {
real alpha;
real beta;
real<lower = 0> sigma;
vector<lower = 0, upper = 1>[N] p;
}
model {
for (n in 1:N) {
real lp[2];
lp[1] = bernoulli_lpmf(0 | p[n])
+ normal_lpdf(Y[n] | alpha + beta * X[n], sigma);
lp[2] = bernoulli_lpmf(1 | p[n])
+ log(1.0e-4);
target += log_sum_exp(lp);
}
alpha ~ normal(0, 10);
beta ~ normal(0, 10);
sigma ~ normal(0, 10);
}
Rコードです。
fit2 <- stan("outlier2.stan",
data = stan_data,
iter = 2000, warmup = 1000, thin = 1)
print(fit2)
ggs(fit2) %>%
ggs_caterpillar(family = "^p")
pの事後分布です。実際に、はずれ値では はずれ値の確率がたかくなっています。
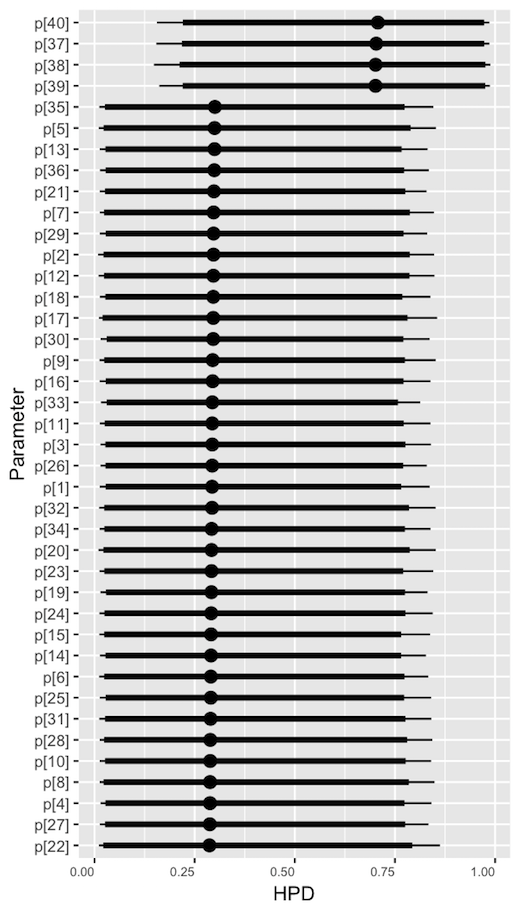
ただし、容易に想像されるとおり、はずれ値が 正しいデータの値にちかくなると、うまくはずれ値と推定されなくなります。その場合はさらに工夫が必要になるでしょう。
タグ:STAn





コメント 0