R: nlme [統計]
nlmeはあまりつかったことがなかった。のでちょっとテストしてみた。
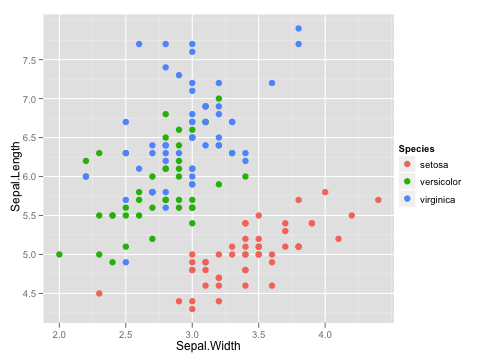
ネタとして適当かどうかわからないが、data(iris)を使用。
べき乗式をあてはめてみる。
結果
結果
ちなみに直線回帰lm()でSpeciesを因子としてモデルに組み込んだ場合は
結果
ネタとして適当かどうかわからないが、data(iris)を使用。
library(ggplot2) data(iris) p <- ggplot(iris, aes(x = Sepal.Width, y = Sepal.Length)) p1 <- p + geom_point(aes(color = Species), size = 3) print(p1)

べき乗式をあてはめてみる。
library(nlme)
sepal.nlme1 <- nlme(Sepal.Length ~ a * Sepal.Width^b,
data = iris,
fixed = a + b ~ 1,
random = a + b ~ 1|Species,
start = c(1, 1), method = "ML")
summary(sepal.nlme1)
結果
Nonlinear mixed-effects model fit by maximum likelihood
Model: Sepal.Length ~ a * Sepal.Width^b
Data: iris
AIC BIC logLik
204.2433 222.3071 -96.12165
Random effects:
Formula: list(a ~ 1, b ~ 1)
Level: Species
Structure: General positive-definite, Log-Cholesky parametrization
StdDev Corr
a 0.62713825 a
b 0.03333759 -0.999
Residual 0.43627681
Fixed effects: a + b ~ 1
Value Std.Error DF t-value p-value
a 3.635606 0.4274668 146 8.505004 0
b 0.433948 0.0583800 146 7.433155 0
Correlation:
a
b -0.773
Standardized Within-Group Residuals:
Min Q1 Med Q3 Max
-2.86192796 -0.58150305 -0.07887569 0.43579445 3.33264845
Number of Observations: 150
Number of Groups: 3
Random effectsにおけるaとbとの相関が-0.999なので、たぶんどちらか一方だけでよい。
sepal.nlme2 <- nlme(Sepal.Length ~ a * Sepal.Width^b,
data = iris,
fixed = a + b ~ 1,
random = a ~ 1|Species,
start = c(1, 1), method = "ML")
summary(sepal.nlme2)
結果
Nonlinear mixed-effects model fit by maximum likelihood
Model: Sepal.Length ~ a * Sepal.Width^b
Data: iris
AIC BIC logLik
200.4443 212.4869 -96.22217
Random effects:
Formula: a ~ 1 | Species
a Residual
StdDev: 0.5005274 0.4365705
Fixed effects: a + b ~ 1
Value Std.Error DF t-value p-value
a 3.662996 0.3687761 146 9.932843 0
b 0.423954 0.0555806 146 7.627739 0
Correlation:
a
b -0.612
Standardized Within-Group Residuals:
Min Q1 Med Q3 Max
-2.80406587 -0.60478687 -0.05863561 0.43341116 3.37435711
Number of Observations: 150
Number of Groups: 3
やはり後者の方がAICが小さい。ちなみに直線回帰lm()でSpeciesを因子としてモデルに組み込んだ場合は
sepal.lm <- lm(Sepal.Length ~ Sepal.Width + Species,
data = iris)
summary(sepal.lm)
AIC(sepal.lm)
結果
> summary(sepal.lm)
Call:
lm(formula = Sepal.Length ~ Sepal.Width + Species, data = iris)
Residuals:
Min 1Q Median 3Q Max
-1.30711 -0.25713 -0.05325 0.19542 1.41253
Coefficients:
Estimate Std. Error t value Pr(>|t|)
(Intercept) 2.2514 0.3698 6.089 9.57e-09 ***
Sepal.Width 0.8036 0.1063 7.557 4.19e-12 ***
Speciesversicolor 1.4587 0.1121 13.012 < 2e-16 ***
Speciesvirginica 1.9468 0.1000 19.465 < 2e-16 ***
---
Signif. codes: 0 ‘***’ 0.001 ‘**’ 0.01 ‘*’ 0.05 ‘.’ 0.1 ‘ ’ 1
Residual standard error: 0.438 on 146 degrees of freedom
Multiple R-squared: 0.7259, Adjusted R-squared: 0.7203
F-statistic: 128.9 on 3 and 146 DF, p-value: < 2.2e-16
> AIC(sepal.lm)
[1] 183.9366
まあ結局、単純なものがよさそう。
タグ:R





コメント 0