誤差のある説明変数 (3) [統計]
誤差のある説明変数のつづき。
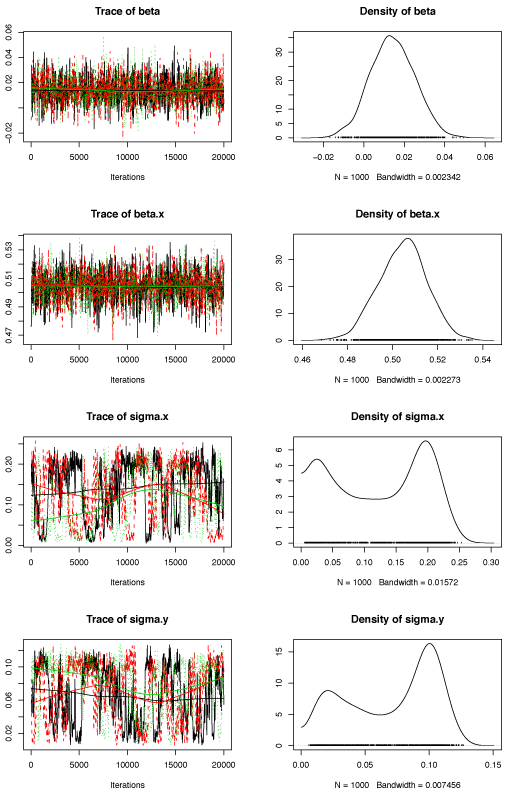
n.iter = 2000, n.thin = 20だと、標準偏差の収束具合が下のように微妙だった。
では、n.iter = 50000, n.thin = 50としてみるとどうだろうか。
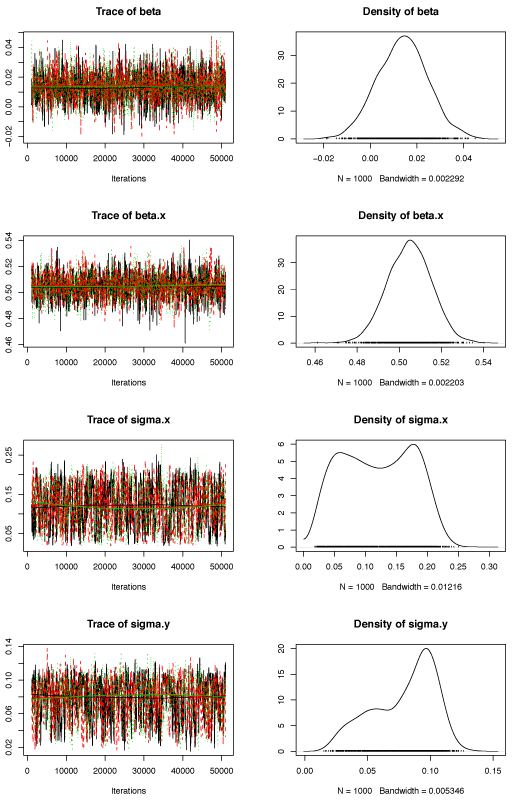
事後分布はやはり二山だが、Traceは収束っぽくなっている。
Gelman-Rubinの収束診断でも1.00になる。
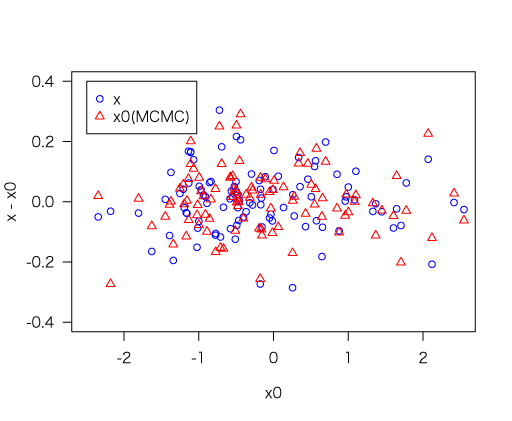
ただし、真のxの値(x0)をうまく推測できているかというと、そうでもない。
Rコード
BUGSコード(x_error.bug)
n.iter = 2000, n.thin = 20だと、標準偏差の収束具合が下のように微妙だった。

では、n.iter = 50000, n.thin = 50としてみるとどうだろうか。

事後分布はやはり二山だが、Traceは収束っぽくなっている。
Gelman-Rubinの収束診断でも1.00になる。
> print(gelman.diag(post[, c("beta", "beta.x", "sigma.x", "sigma.y")]))
Potential scale reduction factors:
Point est. 97.5% quantile
beta 1.00 1.01
beta.x 1.00 1.01
sigma.x 1.00 1.00
sigma.y 1.00 1.00
Multivariate psrf
1.00
ただし、真のxの値(x0)をうまく推測できているかというと、そうでもない。
sum.post <- summary(post)
vars <- varnames(post)
x0.post <- sum.post$statistics[grep("x0", vars), "Mean"]
plot(x0, x - x0, las = 1, col = "blue", pch = 1,
xlim = c(-2.5, 2.5), ylim = c(-0.4, 0.4))
points(x0, x0.post - x0, col = "red", pch = 2)
legend(-2.5, 0.4, c("x", "x0(MCMC)"), col = c("blue", "red"),
pch = c(1, 2))

Rコード
## data
set.seed(1234)
n <- 100 # sample size
sd.x <- 0.1
sd.y <- 0.1
b.x <- 0.5
x0 <- rnorm(n, 0, 1)
x <- x0 + rnorm(n, 0, sd.x)
y <- b.x * x0 + rnorm(n, 0, sd.y)
library(rjags)
model <- jags.model("x_error.bug",
data = list(N = n,
x = x, y = y),
n.chain = 3, n.adapt = 0)
## burn-in
update(model, 1000)
## posterior
post <- coda.samples(model,
variable = c("beta", "beta.x",
"sigma.x", "sigma.y",
"x0", "y0"),
n.iter = 50000, thin = 50)
pdf("x_error_mcmc.pdf", width=512/72, height=768/72,
family = "Helvetica")
plot(post[, c("beta", "beta.x", "sigma.x", "sigma.y")])
dev.off()
BUGSコード(x_error.bug)
var
N, # sample size
x[N], # measured x
y[N], # measured y
x0[N], # real x
y0[N], # real y
## parameters
beta, beta.x,
tau.x, tau.y,
sigma.x, sigma.y
model {
for (i in 1:N) {
y[i] ~ dnorm(y0[i], tau.y);
y0[i] <- beta + beta.x * x0[i];
x[i] ~ dnorm(x0[i], tau.x);
## priors
x0[i] ~ dnorm(0.0, 1.0E-4);
}
## priors
beta ~ dnorm(0.0, 1.0E-4);
beta.x ~ dnorm(0.0, 1.0E-4);
tau.x ~ dgamma(1.0E-3, 1.0E-3);
tau.y ~ dgamma(1.0E-3, 1.0E-3);
sigma.x <- 1/sqrt(tau.x);
sigma.y <- 1/sqrt(tau.y);
}
タグ:MCMC





コメント 0