R: MCMCglmm [統計]
Kuboさんのところや足軽日記さんのところで取り上げられていたMCMCglmmを試してみた。
例によって架空のデータを用意。
まずはglmmMLで。
結果
では、MCMCglmmで。
結果
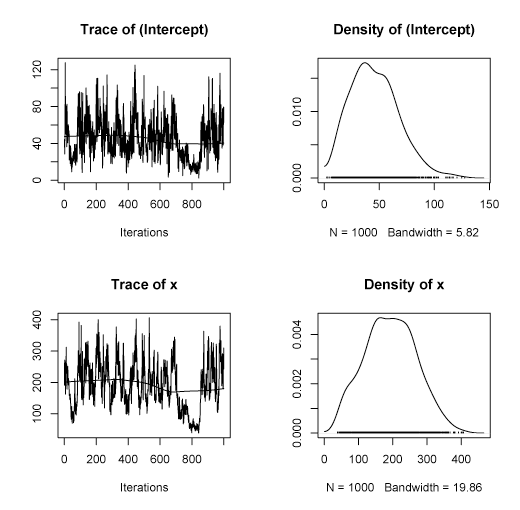
なんか変な値になっている。事前分布の与え方とかがよくわかっていないので(デフォルトだとこの場合エラーになる)、そのあたりで間違っている可能性もあるのだが。
ちなみにJAGSでは。
MCMCglmm_test.bug
結果
例によって架空のデータを用意。
set.seed(11)
i.logit <- function(x) {
exp(x)/(1 + exp(x))
}
n <- 21
block <- as.factor(1:8)
m <- length(block)
x <- seq(-1, 1, length = n)
ranef.sd <- 0.2
ranef <- rnorm(m, 0, ranef.sd)
data <- expand.grid(x = x, block = block)
data$y <- rbinom(n * m, 1,
i.logit(0.7 + 2 * data$x + ranef[data$block]))
まずはglmmMLで。
## glmmML library(glmmML) m.ml <- glmmML(y ~ x, family=binomial, data=data, cluster = block) print(m.ml)
結果
Call: glmmML(formula = y ~ x, family = binomial, data = data, cluster = block)
coef se(coef) z Pr(>|z|)
(Intercept) 0.7693 0.2250 3.420 6.27e-04
x 3.1594 0.4676 6.756 1.42e-11
Scale parameter in mixing distribution: 1.032e-07 gaussian
Std. Error: 0.3634
Residual deviance: 144 on 165 degrees of freedom AIC: 150
では、MCMCglmmで。
## MCMCglmm
library(MCMCglmm)
m.mcmc <- MCMCglmm(y ~ x, random = ~block, family="categorical",
data = data,
prior = list(R = list(n = 1, V = 1),
G = list(G1 = list(n = 1, V = 1))),
nitt = 20000, thin = 10, burnin = 10000)
plot(m.mcmc$Sol)
summary(m.mcmc$Sol)
結果

Error : 外部関数の呼び出し(引数 1) 中に NA/NaN/Inf があります
追加情報: Warning message:
step size truncated due to divergence
Error in glm.fit(x = X, y = Y, weights = weights, start = start, etastart = etastart, :
inner loop 1; cannot correct step size
追加情報: Warning message:
step size truncated due to divergence
Iterations = 1:1000
Thinning interval = 1
Number of chains = 1
Sample size per chain = 1000
1. Empirical mean and standard deviation for each variable,
plus standard error of the mean:
Mean SD Naive SE Time-series SE
(Intercept) 29.44 29.18 0.9226 NA
x 121.83 112.42 3.5552 NA
2. Quantiles for each variable:
2.5% 25% 50% 75% 97.5%
(Intercept) 2.309 4.743 15.10 52.45 94.68
x 12.516 19.943 62.50 230.54 327.38
なんか変な値になっている。事前分布の与え方とかがよくわかっていないので(デフォルトだとこの場合エラーになる)、そのあたりで間違っている可能性もあるのだが。
ちなみにJAGSでは。
## rjags
library(rjags)
model <- jags.model("MCMCglmm_test.bug",
data = list(N = n * m, M = m,
y = data$y,
x = data$x,
b = data$block),
nchain = 3, n.adapt = 5000)
post <- coda.samples(model, c("beta", "beta.x", "sigma"),
n.iter = 10000, thin = 10)
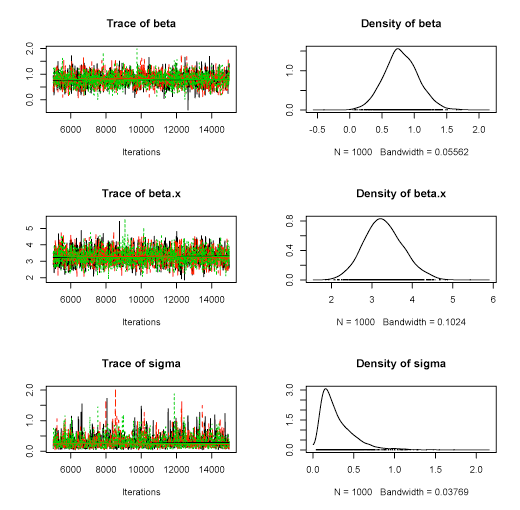
plot(post)
summary(post)
MCMCglmm_test.bug
var
N, # number of data
M, # number of blocks
x[N], y[N], p[N], b[N], e[M],
beta, beta.x, # parameters
tau, sigma; # hyperparameters
model {
for (i in 1:N) {
y[i] ~ dbern(p[i]);
logit(p[i]) <- beta + beta.x * x[i] + e[b[i]];
}
for (j in 1:M) {
e[j] ~ dnorm(0, tau);
}
# priors
beta ~ dnorm(0.0, 1.0E-3);
beta.x ~ dnorm(0.0, 1.0E-3);
tau ~ dgamma(1.0E-2, 1.0E-2);
sigma <- 1/sqrt(tau);
}
結果

Iterations = 5010:15000
Thinning interval = 10
Number of chains = 3
Sample size per chain = 1000
1. Empirical mean and standard deviation for each variable,
plus standard error of the mean:
Mean SD Naive SE Time-series SE
beta 0.7974 0.2660 0.004856 0.004881
beta.x 3.2919 0.4858 0.008870 0.009512
sigma 0.3000 0.2162 0.003947 0.005279
2. Quantiles for each variable:
2.5% 25% 50% 75% 97.5%
beta 0.29698 0.6209 0.7857 0.9696 1.3427
beta.x 2.38036 2.9616 3.2678 3.6035 4.3134
sigma 0.07262 0.1503 0.2394 0.3866 0.8755





コメント 0