R: MCMCpackの並列化 [統計]
ふと、MCMCpackパッケージのMCMCpoisson()関数を並列化してみました。
ポアソン回帰用の模擬データをつくります。
library(MCMCpack) ## Data set.seed(11) N <- 201 x <- seq(0, 1, length = N) y <- rpois(N, exp(3 * x - 1)) d <- data.frame(x = x, y = y)
まずはmclapply()をつかってみます。MCMCpoisson()のseed引数に、要素2つのlistを指定すると、並列化むきというL'Ecuyer random number generatorを使用できます。最初の要素は長さ6のベクトル、2番目の要素はsubstream numberです。詳細はhelp(MCMCpoisson)をご覧ください。
library(parallel)
n.chains <- 3
seeds <- seq.int(from = 11, by = 13, length = 6)
fit <- mclapply(1:n.chains,
function(ch) {
MCMCpoisson(y ~ x, data = d,
burnin = 1000, mcmc = 3000, thin = 3,
verbose = 0, seed = list(seeds, ch))
},
mc.cores = 4)
fit.list <- mcmc.list(fit)
gelman.diag(fit.list)
summary(fit.list)
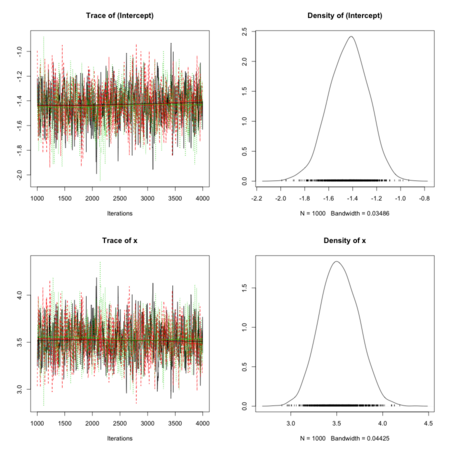
plot(fit.list)
結果です。
> gelman.diag(fit.list)
Potential scale reduction factors:
Point est. Upper C.I.
(Intercept) 1 1.01
x 1 1.01
Multivariate psrf
1
> summary(fit.list)
Iterations = 1001:3998
Thinning interval = 3
Number of chains = 3
Sample size per chain = 1000
1. Empirical mean and standard deviation for each variable,
plus standard error of the mean:
Mean SD Naive SE Time-series SE
(Intercept) -1.436 0.1631 0.002978 0.005010
x 3.527 0.2070 0.003780 0.006333
2. Quantiles for each variable:
2.5% 25% 50% 75% 97.5%
(Intercept) -1.772 -1.548 -1.428 -1.323 -1.126
x 3.138 3.381 3.521 3.670 3.934
つづいて、foreach()で。
##
## foreach
##
library(foreach)
library(doMC)
registerDoMC(4)
fit2 <- foreach(ch = 1:n.chains) %dopar% {
MCMCpoisson(y ~ x, data = d,
burnin = 1000, mcmc = 3000, thin = 3,
verbose = 0, seed = list(seeds, ch))
}
fit2.list <- mcmc.list(fit2)
gelman.diag(fit2.list)
summary(fit2.list)
結果です。
> gelman.diag(fit2.list)
Potential scale reduction factors:
Point est. Upper C.I.
(Intercept) 1 1.01
x 1 1.01
Multivariate psrf
1
> summary(fit2.list)
Iterations = 1001:3998
Thinning interval = 3
Number of chains = 3
Sample size per chain = 1000
1. Empirical mean and standard deviation for each variable,
plus standard error of the mean:
Mean SD Naive SE Time-series SE
(Intercept) -1.436 0.1631 0.002978 0.005010
x 3.527 0.2070 0.003780 0.006333
2. Quantiles for each variable:
2.5% 25% 50% 75% 97.5%
(Intercept) -1.772 -1.548 -1.428 -1.323 -1.126
x 3.138 3.381 3.521 3.670 3.934
乱数のseedを指定してあるので、結果は一致します。
ただ、これくらいの計算量だと並列化の御利益はありません。





コメント 0