2次元でのStanによるCARモデルのテスト [統計]
berobero11さんのStanによるCARモデルを2次元でためしてみたテストです。
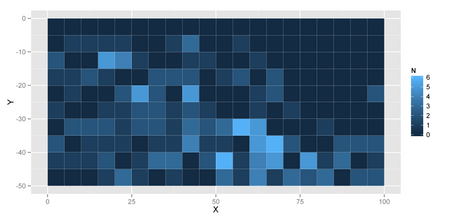
使用したデータは、銀閣寺山国有林(京都市左京区)で取得したアラカシという木の分布データです。50m×100mの調査地を5m×5mのメッシュに分割して、それぞれのメッシュに含まれるアラカシの個体数(株数)を測定しました。方法の詳細は、伊東(2007)をご覧ください。1993年のデータを使用しました。
データ→Qglauca.csv
図示してみると以下のようになります。
## http://cse.ffpri.affrc.go.jp/hiroki/stat/Qglauca.csv qg <- read.csv("Qglauca.csv") ## Plot library(ggplot2) ggplot(qg) + geom_tile(aes(x = X + 2.5, y = Y - 2.5, fill = N)) + xlab("X") + ylab("Y")
まずはOpenBUGSでCARモデルにあてはめをしてみます。
## Conditional Autoregression Model
## adjacent plots
adj <- c(2, 11, # 1
c(sapply(2:9, function(i) i + c(-1, 1, 10))), # 2--9
9, 20, # 10
c(sapply(seq(0, 170, 10),
function(j) j + c(1, 12, 21,
c(sapply(2:9,
function(i) i + c(0, 9, 11, 20))),
10, 19, 30))), # 10--190
181, 192, # 191
c(sapply(2:9, function(i) i + c(180, 189, 191))), # 192--199
190, 199) # 200
num.adj <- c(2, rep(3, 8), 2,
rep(c(3, rep(4, 8), 3), 18),
2, rep(3, 8), 2)
weights <- rep(1, length(adj))
## OpenBUGS
library(R2OpenBUGS)
## OS X (Darwin) なら/Applications/Wine.appを使用
## Linuxなら/usr/local/bin/OpenBUGSを使用
uname <- system("uname", intern = TRUE)
if (uname == "Darwin") {
Sys.setenv(DYLD_FALLBACK_LIBRARY_PATH="/usr/x11/lib")
kOpenBUGS <- paste(Sys.getenv("HOME"),
".wine/drive_c/Program\ Files",
"OpenBUGS/OpenBUGS322/OpenBUGS.exe", sep = "/")
kWine <- "/Applications/Wine.app/Contents/Resources/bin/wine"
kWinePath <- "/Applications/Wine.app/Contents/Resources/bin/winepath"
useWine <- TRUE
debug <- TRUE
} else if (uname == "Linux") {
kOpenBUGS <- "/usr/local/bin/OpenBUGS"
kWine <- NULL
kWinePath <- NULL
useWine <- FALSE
debug <- FALSE
}
## BUGS model
bugs.model <- "
var
## data
N_site, # number of subquadrats
Y[N_site], # stem number of Q.glauca
AdjNum[N_site], # number of adjacent plots
N_Adj, # length of Adj[]
Adj[N_Adj], # adjacent plots
Weights[N_Adj], # weights
## parameters
beta, # intercept
lamdba[N_site], # mean number of individuals
r[N_site] # spatial error
tau,
sigma;
model {
for (i in 1:N_site) {
Y[i] ~ dpois(lambda[i]);
log(lambda[i]) <- beta + r[i];
}
r[1:N_site] ~ car.normal(Adj[], Weights[], AdjNum[], tau);
beta ~ dnorm(0, 1.0E-4);
tau <- 1 / (sigma * sigma);
sigma ~ dunif(0, 1.0E+4);
}"
model.file <- "Qglauca_bugs.txt"
cat(bugs.model, file = model.file)
## parameters to save
params <- c("beta", "sigma", "lambda")
iter <- 7000
burnin <- 2000
thin <- 5
##
data <- list(N_site = nrow(qg),
Y = qg$N,
AdjNum = num.adj,
N_Adj = length(num.adj),
Adj = adj,
Weights = weights)
inits <- list(list(beta = 0, sigma = 1),
list(beta = 1, sigma = 2),
list(beta = 3, sigma = 0.1))
samp <- bugs(data = data,
model.file = model.file,
inits = inits,
parameters = params,
n.chains = 3,
n.iter = iter, n.burnin = burnin, n.thin = thin,
OpenBUGS = kOpenBUGS,
useWINE = useWine,
WINE = kWine,
WINEPATH = kWinePath,
debug = debug)
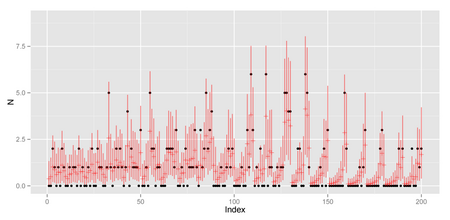
結果
> print(samp, digits = 3)
Inference for Bugs model at "Qglauca_bugs.txt",
Current: 3 chains, each with 7000 iterations (first 2000 discarded), n.thin = 5
Cumulative: n.sims = 15000 iterations saved
mean sd 2.5% 25% 50% 75% 97.5% Rhat n.eff
beta -0.528 0.130 -0.800 -0.612 -0.522 -0.438 -0.285 1.001 5500
sigma 1.168 0.175 0.848 1.048 1.158 1.280 1.537 1.001 15000
[...]
deviance 434.622 15.764 404.300 424.000 434.300 444.900 466.600 1.001 6400
For each parameter, n.eff is a crude measure of effective sample size,
and Rhat is the potential scale reduction factor (at convergence, Rhat=1).
DIC info (using the rule, pD = Dbar-Dhat)
pD = 2.4 and DIC = 437.0
DIC is an estimate of expected predictive error (lower deviance is better).
library(ggplot2)
N_site <- nrow(qg)
N = qg$N
ggplot(data.frame(x = 1:N_site,
y = N,
ymean = samp$mean$lambda,
ymax = apply(samp$sims.list$lambda, 2, quantile, 0.975),
ymin = apply(samp$sims.list$lambda, 2, quantile, 0.025))) +
geom_point(aes(x = x, y = y)) +
geom_pointrange(aes(x = x, y = ymean, ymax = ymax, ymin = ymin),
colour = "red", alpha = 0.5, shape = 3) +
xlab("Index") + ylab("N") + ylim(0, 9)
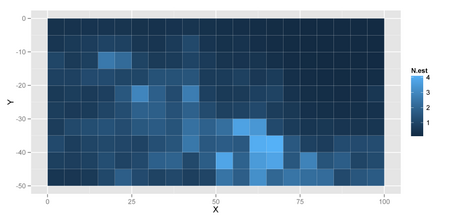
ggplot(data.frame(x = qg$X, y = qg$Y,
N.est = samp$mean$lambda)) +
geom_tile(aes(x = x + 2.5, y = y - 2.5, fill = N.est)) +
xlab("X") + ylab("Y")
つづいて、Stanで。Stanのコードはほぼberobero11さんのコードそのままです。CAR.stanというファイル名で保存。
/* CAR.stan CAR model created by @berobero11 http://heartruptcy.blog.fc2.com/blog-entry-141.html */ data { int<-lower=1> N_site; int<-lower=1> N_Adj; int<-lower=0> Y[N_site]; int<-lower=1> Adj[N_Adj]; real<-lower=0> Weight[N_Adj]; int<-lower=1> Offset[N_site, 2]; } parameters { real r0[N_site-1]; real beta; real<-lower=0> sigma; } transformed parameters { real r[N_site]; real s[N_site]; real mu[N_site]; for (j in 1:(N_site-1)) r[j] <- r0[j]; r[N_site] <- -sum(r0); for (j in 1:N_site) { real sum_x; real sum_w; sum_x <- 0.0; sum_w <- 0.0; for (i in Offset[j, 1]:Offset[j, 2]) { sum_x <- sum_x + Weight[i] * r[Adj[i]]; sum_w <- sum_w + Weight[i]; } mu[j] <- sum_x / sum_w; s[j] <- sigma / sqrt(sum_w); } } model { for (j in 1:N_site) { Y[j] ~ poisson_log(beta + r[j]); r[j] ~ normal(mu[j], s[j]); } beta ~ normal(0, 1.0e+2); sigma ~ uniform(0, 1.0e+4); } generated quantities { real<-lower=0> Y_mean[N_site]; for (j in 1:N_site) Y_mean[j] <- exp(beta + r[j]); }
OS X MavericksでR 3.1.0だと、RStanがこけるので、CmdStanを使用しました。
## データを用意
qg <- read.csv("Qglauca.csv")
adj <- c(2, 11, # 1
c(sapply(2:9, function(i) i + c(-1, 1, 10))), # 2--9
9, 20, # 10
c(sapply(seq(0, 170, 10),
function(j) j + c(1, 12, 21,
c(sapply(2:9,
function(i) i + c(0, 9, 11, 20))),
10, 19, 30))), # 10--190
181, 192, # 191
c(sapply(2:9, function(i) i + c(180, 189, 191))), # 192--199
190, 199) # 200
num.adj <- c(2, rep(3, 8), 2,
rep(c(3, rep(4, 8), 3), 18),
2, rep(3, 8), 2)
weights <- rep(1, length(adj))
N_site <- nrow(qg)
offset2 <- offset1 <- rep(NA, N_site)
offset1[1] <- 1
for (i in 2:N_site) {
offset1[i] <- 1 + sum(num.adj[1:(i - 1)])
}
for (i in 1:(N_site - 1)) {
offset2[i] <- offset1[i + 1] - 1
}
offset2[N_site] <- length(adj)
Offset <- cbind(offset1, offset2)
colnames(Offset) <- NULL
Y <- qg$N
Adj <- adj
N_Adj <- length(Adj)
Weight <- weights
dump(c("Y", "N_site", "N_Adj", "Adj", "Weight", "Offset"),
file = "Qglauca_dump.R")
CAR.stanをコンパイルしておいて、以下のシェルスクリプトを実行。
#!/bin/sh
ITER=5000
WARMUP=2000
THIN=5
DATA=Qglauca_dump.R
OUT=Qglauca
./CAR sample num_samples=${ITER} num_warmup=${WARMUP} thin=${THIN} \
id=1 data file=${DATA} random seed=123 output file=${OUT}_1.csv \
refresh=1000 &
./CAR sample num_samples=${ITER} num_warmup=${WARMUP} thin=${THIN} \
id=2 data file=${DATA} random seed=345 output file=${OUT}_2.csv \
refresh=1000 &
./CAR sample num_samples=${ITER} num_warmup=${WARMUP} thin=${THIN} \
id=3 data file=${DATA} random seed=567 output file=${OUT}_3.csv \
refresh=1000 &
結果のとりこみ
library(coda)
fit <- list()
for (i in 1:3) {
fit[[i]] <- as.mcmc(read.csv(paste("Qglauca_", i, ".csv", sep = ""),
comment = "#"))
}
fit.mcmc.list <- as.mcmc.list(fit)
summary(fit.mcmc.list[, c("beta", "sigma")])
gelman.diag(fit.mcmc.list[, c("beta", "sigma")],
multivariate = FALSE)
params <- colnames(fit[[1]])
pos.Y_mean <- match("Y_mean.1", params)
Y_mean <- fit.mcmc.list[, pos.Y_mean:(pos.Y_mean + N_site - 1)]
summary.Y_mean <- summary(Y_mean)
結果の表示
> summary(fit.mcmc.list[, c("beta", "sigma")])
Iterations = 1:1000
Thinning interval = 1
Number of chains = 3
Sample size per chain = 1000
1. Empirical mean and standard deviation for each variable,
plus standard error of the mean:
Mean SD Naive SE Time-series SE
beta -0.4753 0.1344 0.002454 0.005060
sigma 0.3728 0.1036 0.001891 0.006123
2. Quantiles for each variable:
2.5% 25% 50% 75% 97.5%
beta -0.7532 -0.5609 -0.4685 -0.3853 -0.2306
sigma 0.2145 0.3006 0.3571 0.4264 0.6237
> gelman.diag(fit.mcmc.list[, c("beta", "sigma")],
+ multivariate = FALSE)
Potential scale reduction factors:
Point est. Upper C.I.
beta 1 1.01
sigma 1 1.02
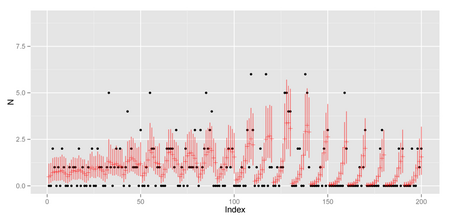
グラフを表示
library(ggplot2)
ggplot(data.frame(x = 1:N_site,
y = Y,
ymean = summary.Y_mean$statistic[, "Mean"],
ymax = summary.Y_mean$quantiles[, "97.5%"],
ymin = summary.Y_mean$quantiles[, "2.5%"])) +
geom_point(aes(x = x, y = y)) +
geom_pointrange(aes(x = x, y = ymean, ymax = ymax, ymin = ymin),
colour = "red", alpha = 0.5, shape = 3) +
xlab("Index") + ylab("N") + ylim(0, 9)
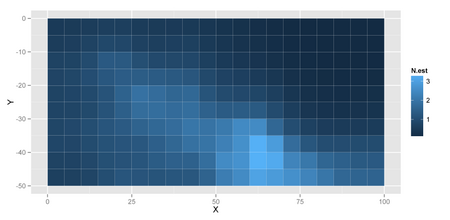
ggplot(data.frame(x = qg$X, y = qg$Y,
N.est = summary.Y_mean$statistic[, "Mean"])) +
geom_tile(aes(x = x + 2.5, y = y - 2.5, fill = N.est)) +
xlab("X") + ylab("Y")
どうもStanの方が空間誤差の分散が小さく推定されるようです。 と、ここまで来たところで まだ原因は追及していません。どこか間違えているかも。





コメント 0