MAGROの推定結果を検証する [統計]
MAGROの推定結果を他のソフトによるものと比較してみる。
まず、bugs-examples/vol1/blockerの結果をJAGSとMAGROとで比較してみる。各パラメータの事後分布の平均の分布を2000回のモンテカルロシミュレーションで推定した。
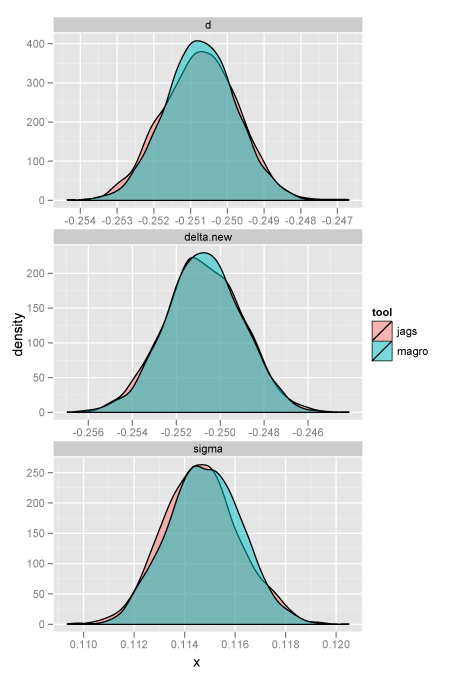
ほぼ同じ結果となっている。
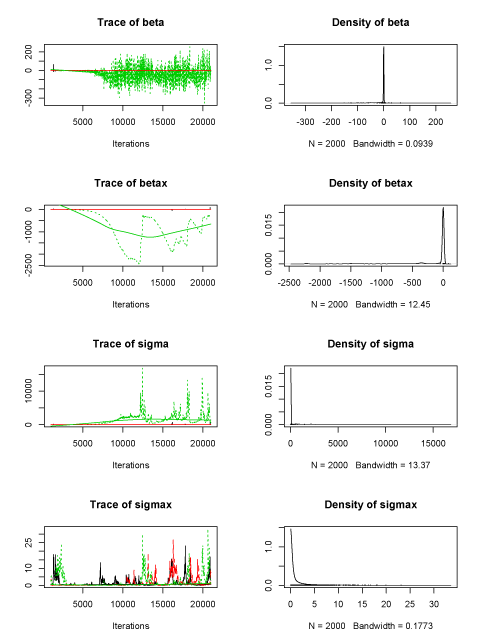
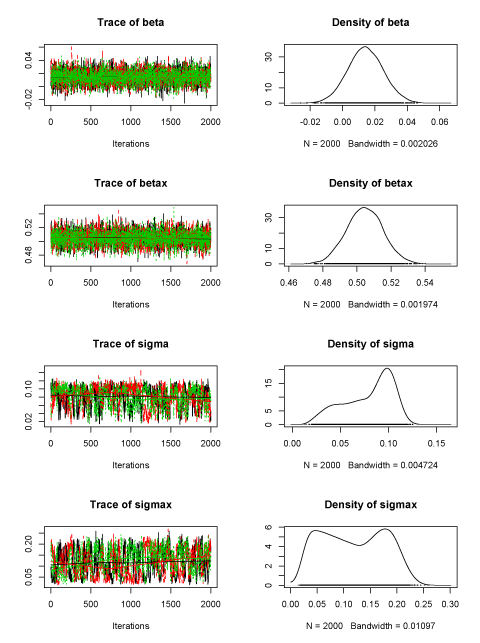
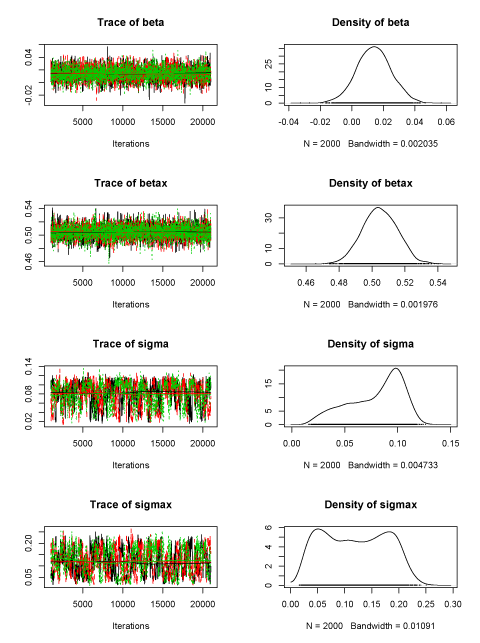
ただし、あまりうまく収束しないモデルではMAGROがほんとうに収束しないということもあるようだ。下は、誤差のあるX変数のモデルの推定結果を比較したもの。
MAGRO
WinBUGS
JAGS
使用したコード
モデル
まず、bugs-examples/vol1/blockerの結果をJAGSとMAGROとで比較してみる。各パラメータの事後分布の平均の分布を2000回のモンテカルロシミュレーションで推定した。

ほぼ同じ結果となっている。
ただし、あまりうまく収束しないモデルではMAGROがほんとうに収束しないということもあるようだ。下は、誤差のあるX変数のモデルの推定結果を比較したもの。
MAGRO

WinBUGS

JAGS

使用したコード
###
### Compare MAGRO, JAGS and WinBUGS
###
library(rmagro)
library(rjags)
library(R2WinBUGS)
## data
set.seed(1234)
n <- 100
beta <- 0
beta.x <- 0.5
sd.x <- 0.1
sd.y <- 0.1
x0 <- rnorm(n, 0, 1)
x <- x0 + rnorm(n, 0, sd.x)
y <- beta + beta.x * x0 + rnorm(n, 0, sd.y)
## model
model.file <- "test3.bug"
## inits
init <- list(beta = 0, betax = 0,
taux = 1, tau = 1)
## parameters to monitor
parms <- c("beta", "betax", "sigma", "sigmax")
## MAGRO
post.magro <- magro.samples(model = model.file,
n.iter = 21000,
n.burnin = 1000,
list.data = list(N = n,
x1 = x, y1 = y),
inits = init,
thin = 10,
n.chain = 3,
monitor = parms)
## JAGS
model <- jags.model(model.file,
data = list(N = n, x1 = x, y1 = y),
inits = list(init, init, init),
n.chains = 3, n.adapt = 0)
update(model, 1000)
post.jags <- coda.samples(model, parms,
n.iter = 20000, thin = 10)
## WinBUGS
WINE <- "/Applications/Darwine/Wine.bundle/Contents/bin/wine"
WINEPATH <- "/Applications/Darwine/Wine.bundle/Contents/bin/winepath"
bugs.dir <- paste(Sys.getenv("HOME"),
".wine/drive_c/Program\ Files/WinBUGS14", sep = "/")
samp <- bugs(data = list(N = n, x1 = x, y1 = y),
inits = list(init, init, init), parms,
model.file,
n.chains = 3, n.iter = 21000, n.burnin = 1000,
n.thin = 10, debug = FALSE,
bugs.directory = bugs.dir,
working.directory = getwd(),
useWINE = TRUE, WINE = WINE, WINEPATH = WINEPATH)
post.winbugs <- mcmc.list(lapply(1:(samp$n.chain),
function(chain)
as.mcmc(samp$sims.array[,chain,])))
モデル
## model 1
var
N, # sample size
x1[N], # measured x
y1[N], # measured y
x0[N], # real x
muy[N],
## parameters
beta, betax,
taux, tau,
sigmax, sigma
model {
for (i in 1:N) {
y1[i] ~ dnorm(muy[i], tau);
muy[i] <- beta + betax * x0[i];
x1[i] ~ dnorm(x0[i], taux);
## prior
x0[i] ~ dnorm(0.0, 1.0E-4);
}
## priors
beta ~ dnorm(0.0, 1.0E-4);
betax ~ dnorm(0.0, 1.0E-4);
taux ~ dgamma(1.0E-3, 1.0E-3);
tau ~ dgamma(1.0E-3, 1.0E-3);
sigmax <- 1/sqrt(taux);
sigma <- 1/sqrt(tau);
}





コメント 0