またもやXの誤差 [統計]
誤差を含んだXでの回帰をもうちょっとかんがえてみる。今回は2000回計算を繰り返して傾きの値の分布を推定する。
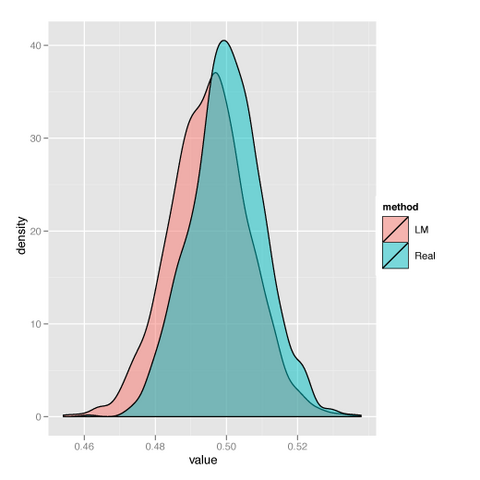
実際のXの値で回帰したときと、誤差を含んだXの値で直線回帰したときの傾きの値。
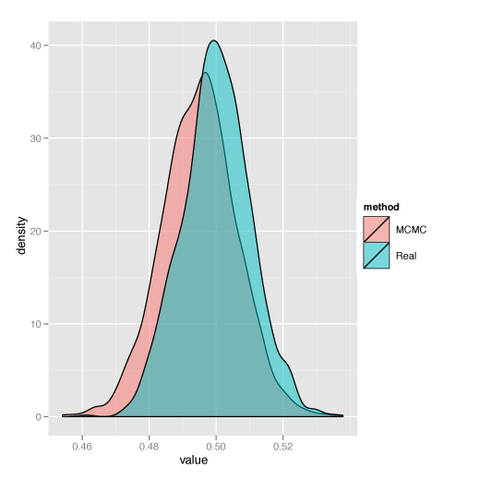
実際のXの値で回帰したときと、誤差を含んだXの値をつかって下のモデルでMCMC推定したときの傾きの値。
なんだかほとんどおなじになっている。
直線回帰のコード
MCMCのコード
JAGS_R.RはRからJAGSを呼び出す自作コード。rjagsのない環境でつかうのでこれを使っている。

実際のXの値で回帰したときと、誤差を含んだXの値で直線回帰したときの傾きの値。

実際のXの値で回帰したときと、誤差を含んだXの値をつかって下のモデルでMCMC推定したときの傾きの値。
なんだかほとんどおなじになっている。
直線回帰のコード
###
### error in x
###
## data
set.seed(1234)
test1 <- function(beta = 0, beta.x = 0.5,
n = 100,
sd.x = 0.1, sd.y = 0.1) {
x0 <- rnorm(n, 0, 1)
x <- x0 + rnorm(n, 0, sd.x)
y <- beta + beta.x * x0 + rnorm(n, 0, sd.y)
c0 <- lm(y ~ x0)$coefficients["x0"]
c1 <- lm(y ~ x)$coefficients["x"]
c(c0, c1)
}
t1 <- replicate(2000, test1(beta = 0, beta.x = b.x,
n = n,
sd.x = sd.x, sd.y = sd.y))
save(t1, file = "t1.RData")
pdf("x_error_lm.pdf", family = "Helvetica", pointsize = 12,
width = 640/72, height = 480/72)
plot(density(t1[1,]), col = "black", las = 1, main = "density")
lines(density(t1[2,]), col = "red")
dev.off()
MCMCのコード
###
### error in x
### MCMC
###
source("~/Documents/RData/JAGS_R.R")
model.file <- "model_mcmc1.bug"
jags <- system("which jags", intern = TRUE)
## model
mod <- "## model 1
var
N, # sample size
x[N], # measured x
y[N], # measured y
x0[N], # real x
y0[N], # real y
## parameters
beta, beta.x,
tau.x, tau.y,
sigma.x, sigma.y
model {
for (i in 1:N) {
y[i] ~ dnorm(y0[i], tau.y);
y0[i] <- beta + beta.x * x0[i];
x[i] ~ dnorm(x0[i], tau.x);
## prior
x0[i] ~ dnorm(0.0, 1.0E-4);
}
## priors
beta ~ dnorm(0.0, 1.0E-4);
beta.x ~ dnorm(0.0, 1.0E-4);
tau.x ~ dgamma(1.0E-3, 1.0E-3);
tau.y ~ dgamma(1.0E-3, 1.0E-3);
sigma.x <- 1/sqrt(tau.x);
sigma.y <- 1/sqrt(tau.y);
}
"
write(mod, file = model.file)
## data
set.seed(1234)
n <- 100 # sample size
sd.x <- 0.1
sd.y <- 0.1
b.x <- 0.5
test2 <- function(beta = 0, beta.x = 0.5,
n = 100,
sd.x = 0.1, sd.y = 0.1) {
x0 <- rnorm(n, 0, 1)
x <- x0 + rnorm(n, 0, sd.x)
y <- beta + beta.x * x0 + rnorm(n, 0, sd.y)
init1 <- list(beta = 0, beta.x = 0,
tau.x = 1, tau.y = 1)
init2 <- list(beta = -10, beta.x = -10,
tau.x = 10, tau.y = 10)
init3 <- list(beta = 10, beta.x = 10,
tau.x = 0.01, tau.y = 0.01)
post <- jags.run(data = list(N = n, x = x, y = y),
inits = list(init1, init2, init3),
parameters.to.save = c("beta", "beta.x"),
model.file = model.file,
data.file = "mcmc1-data.R",
init.file = "mcmc1-init.R",
cmd.file = "mcmc1.cmd",
out = "mcmc1CODA",
jags = jags,
n.chains = 3,
n.iter = 51000,
n.burnin = 1000,
n.thin = 50)
post.sum <- summary(post)
c0 <- lm(y ~ x0)$coefficients["x0"]
c1 <- post.sum$statistics["beta.x", "Mean"]
c(c0, c1)
}
mcmc1 <- replicate(2000, test2(beta = 0, beta.x = b.x,
n = n,
sd.x = sd.x, sd.y = sd.y))
save(mcmc1, file = "mcmc1.RData")
JAGS_R.RはRからJAGSを呼び出す自作コード。rjagsのない環境でつかうのでこれを使っている。





コメント 0