累積ロジットモデルのベイズ推定(2) [統計]
前回のつづき。今度はランダム効果の入ったデータを推定してみる。
まずはデータを用意。
# inverse logit
invlogit <- function(x) exp(x)/(1+exp(x))
set.seed(12345)
n.rank <- 5
plot <- 1:20
n.plot <- length(plot)
time <- 1:5
n.time <- length(time)
n <- n.plot * n.time
data <- expand.grid(plot = plot, time = time)
slope <- rgamma(n.plot, shape = 15^2/5^2, rate = 15/5^2)
data$slope <- rep(slope, n.time)
ranef.plot <- rnorm(n.plot, 0, 1)
ranef.time <- rnorm(n.time, 0, 1)
logit.c1 <- 7.5 - 0.5 * slope +
ranef.plot[data$plot] + ranef.time[data$time]
data$c1 <- rbinom(n, n.rank - 1, invlogit(logit.c1)) + 1
累積データを作成して、JAGSにかける。
cum <- matrix(NA, nrow = n.plot * n.time, ncol = n.rank - 1)
cum[,1] <- as.numeric(data$c1 >= 2)
cum[,2] <- as.numeric(data$c1 >= 3)
cum[,3] <- as.numeric(data$c1 >= 4)
cum[,4] <- as.numeric(data$c1 >= 5)
library(rjags)
model <- jags.model("cumlogit_MCMC2.bug",
data = list(N = n, M = n.rank - 1,
K = n.plot, L = n.time,
s = data$slope, c = cum,
plot = data$plot, time = data$time),
nchain = 3, n.adapt = 10000)
post <- coda.samples(model,
variable = c("b", "beta", "sigma.p", "sigma.t"),
n.iter = 20000, thin = 20)
BUGSコード
var
N, # number of data
M, # number of ranks
K, # number of plots
L, # number of mesurements
beta,
b[M],
c[N, M],
s[N],
plot[N], e.plot[K],
time[N], e.time[L];
model {
for (j in 1:M) {
for (i in 1:N) {
c[i, j] ~ dbern(p[i, j]);
logit(p[i, j]) <- b[j] + beta * s[i] +
e.plot[plot[i]] + e.time[time[i]];
}
b[j] ~ dnorm(0.0, 1.0E-4);
}
for (i in 1:K) {
e.plot[i] ~ dnorm(0, tau.p);
}
for (i in 1:L) {
e.time[i] ~ dnorm(0, tau.t);
}
beta ~ dnorm(0.0, 1.0E-4);
tau.p ~ dgamma(1.0E-3, 1.0E-3);
tau.t ~ dgamma(1.0E-3, 1.0E-3);
sigma.p <- 1/sqrt(tau.p);
sigma.t <- 1/sqrt(tau.t);
}
結果
> summary(post)
Iterations = 10020:30000
Thinning interval = 20
Number of chains = 3
Sample size per chain = 1000
1. Empirical mean and standard deviation for each variable,
plus standard error of the mean:
Mean SD Naive SE Time-series SE
b[1] 14.4082 2.6237 0.047902 0.195126
b[2] 11.6440 2.4110 0.044018 0.180754
b[3] 10.2122 2.3343 0.042619 0.172398
b[4] 7.4226 2.1665 0.039554 0.152169
beta -0.7306 0.1376 0.002513 0.009946
sigma.p 2.0650 0.5855 0.010690 0.019606
sigma.t 1.6044 0.8677 0.015841 0.021022
2. Quantiles for each variable:
2.5% 25% 50% 75% 97.5%
b[1] 9.8577 12.6381 14.235 15.9341 19.9900
b[2] 7.4117 10.0158 11.449 13.0504 16.8387
b[3] 6.1724 8.5996 10.039 11.5374 15.2372
b[4] 3.5728 5.9820 7.287 8.6834 12.0245
beta -1.0236 -0.8163 -0.720 -0.6385 -0.4859
sigma.p 1.1528 1.6539 1.988 2.3900 3.4480
sigma.t 0.6625 1.0580 1.399 1.8622 3.8623
同じデータをdrmで解析してみる。
library(drm)
fit <- drm(c1 ~ slope + cluster(plot) + Time(time),
data = data,
start = c(0, 1, 2, 3, -1),
print.level = 0)
結果
> summary(fit)
Call:
drm(formula = c1 ~ slope + cluster(plot) + Time(time), data = data,
start = c(0, 1, 2, 3, -1), print.level = 0)
Coefficients:
Value Std. Error z value
(Intercept)1 -8.707243 1.09793339 -7.930575
(Intercept)2 -7.003733 0.94661067 -7.398747
(Intercept)3 -6.134059 0.88513704 -6.930067
(Intercept)4 -4.475916 0.79272635 -5.646230
slope -0.444208 0.05900017 -7.528927
Residual deviance: 216.881 AIC: 226.881
Convergence code 1 in 32 iterations (See help(nlm) for details)
Correlation of Coefficients:
(Intercept)1 (Intercept)2 (Intercept)3 (Intercept)4
(Intercept)2 0.948
(Intercept)3 0.933 0.966
(Intercept)4 0.892 0.909 0.924
slope 0.957 0.957 0.953 0.921
なんだか微妙に結果が違うが、何が原因なのだろう? 推定法の違いによるものなのか、どっか間違えているのか。切片の符号が逆なのは、モデルでの符号の与え方の違いによるものなので、それは問題ない。
データに期待値の線を重ねてプロットしてみる。
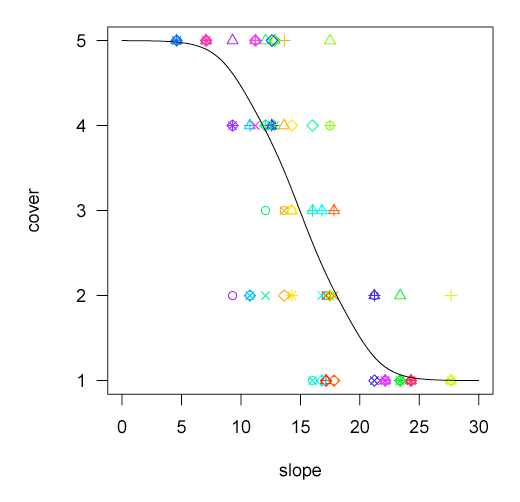
ベイズ推定によるもの
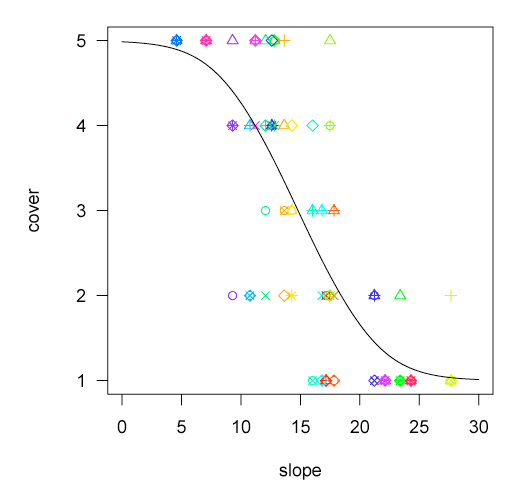
drmによるもの
タグ:累積ロジットモデル





コメント 0