多重共線性とか潜在変数とか共分散構造分析とか(3) [統計]
さらに承前。もうちょっと複雑なモデル。
まずはデータ。
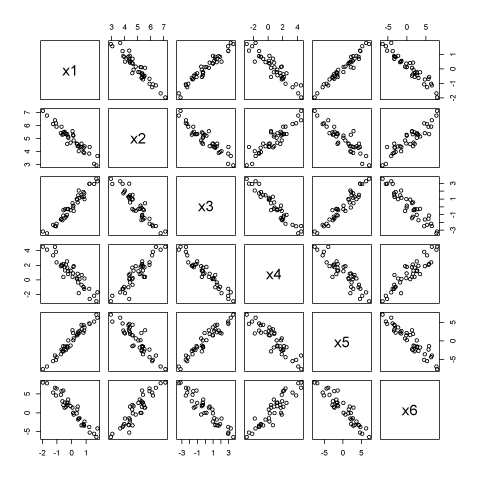
set.seed(123) N <- 40 x1 <- rnorm(N, 0, 1) x2 <- 5 - x1 + rnorm(N, 0, 0.3) x3 <- 2 * x1 + rnorm(N, 0, 0.5) x4 <- 1 - x3 + rnorm(N, 0, 0.5) x5 <- 4 * x1 + rnorm(N, 0, 0.8) x6 <- 1 - 4 * x1 + rnorm(N, 0, 1.2) data <- cbind(x1, x2, x3, x4, x5, x6) pairs(data)

こういうモデル。beta1の入るべきところを0で固定。lambda11を正に固定。
var
N, # number of data
x1[N], x2[N], x3[N], x4[N], x5[N], x6[N], # data
xi1[N], eta1[N], # latent variables
lambda11, lambda21, lambda31, lambda41, # parameters
kappa51, kappa61, mu,
mu1[N], mu2[N], mu3[N], mu4[N], mu5[N], mu6[N],
tau1, tau2, tau3, tau4, tau5, tau6,
sigma1, sigma2, sigma3, sigma4, sigma5, sigma6
model {
for (i in 1:N) {
x1[i] ~ dnorm(mu1[i], tau1);
mu1[i] <- 0 + lambda11 * xi1[i];
x2[i] ~ dnorm(mu2[i], tau2);
mu2[i] <- beta2 + lambda21 * xi1[i];
x3[i] ~ dnorm(mu3[i], tau3);
mu3[i] <- beta3 + lambda31 * xi1[i];
x4[i] ~ dnorm(mu4[i], tau4);
mu4[i] <- beta4 + lambda41 * xi1[i];
x5[i] ~ dnorm(mu5[i], tau5);
mu5[i] <- beta5 + kappa51 * eta1[i];
x6[i] ~ dnorm(mu6[i], tau6);
mu6[i] <- beta6 + kappa61 * eta1[i];
eta1[i] ~ dnorm(xi1[i], 1);
xi1[i] ~ dnorm(mu, 1);
}
lambda11 ~ dgamma(1.0E-3, 1.0E-3);
lambda21 ~ dnorm(0, 1.0E-6);
lambda31 ~ dnorm(0, 1.0E-6);
lambda41 ~ dnorm(0, 1.0E-6);
kappa51 ~ dnorm(0, 1.0E-6);
kappa61 ~ dnorm(0, 1.0E-6);
mu ~ dnorm(0, 1.0E-6);
# beta1 ~ dnorm(0, 1.0E-6);
beta2 ~ dnorm(0, 1.0E-6);
beta3 ~ dnorm(0, 1.0E-6);
beta4 ~ dnorm(0, 1.0E-6);
beta5 ~ dnorm(0, 1.0E-6);
beta6 ~ dnorm(0, 1.0E-6);
tau1 ~ dgamma(1.0E-3, 1.0E-3);
tau2 ~ dgamma(1.0E-3, 1.0E-3);
tau3 ~ dgamma(1.0E-3, 1.0E-3);
tau4 ~ dgamma(1.0E-3, 1.0E-3);
tau5 ~ dgamma(1.0E-3, 1.0E-3);
tau6 ~ dgamma(1.0E-3, 1.0E-3);
sigma1 <- 1/sqrt(tau1);
sigma2 <- 1/sqrt(tau2);
sigma3 <- 1/sqrt(tau3);
sigma4 <- 1/sqrt(tau4);
sigma5 <- 1/sqrt(tau5);
sigma6 <- 1/sqrt(tau6);
}
ひきつづきrjagsを使用。
library(rjags)
mcmc.data <- list(N = N, x1 = x1, x2 = x2, x3 = x3,
x4 = x4, x5 = x5, x6 = x6)
mcmc.model <- jags.model("sem.bug", mcmc.data,
nchain = 3,
n.adapt = 10000)
vars <- c("lambda11", "lambda21", "lambda31",
"lambda41", "kappa51", "kappa61",
# "beta1",
"beta2", "beta3",
"beta4", "beta5", "beta6",
"sigma1", "sigma2", "sigma3", "sigma4",
"sigma5", "sigma6", "mu")
mcmc.samples <- coda.samples(mcmc.model, vars,
n.iter = 30000, thin = 30)
結果
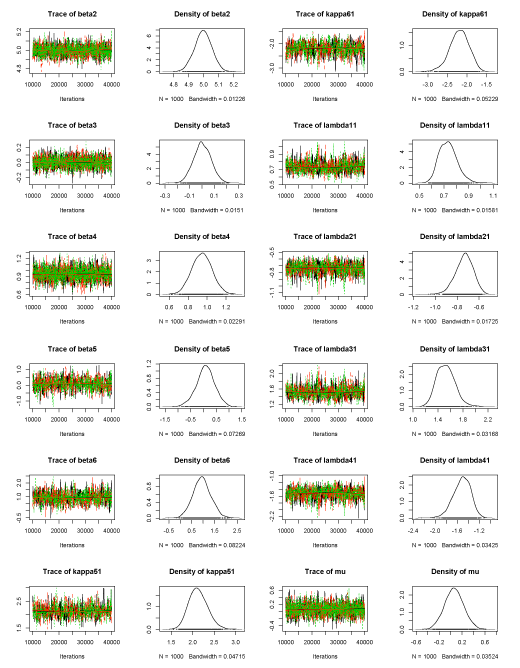
Iterations = 10030:40000
Thinning interval = 30
Number of chains = 3
Sample size per chain = 1000
1. Empirical mean and standard deviation for each variable,
plus standard error of the mean:
Mean SD Naive SE Time-series SE
beta2 4.996584 0.05814 0.0010616 0.0011823
beta3 0.003045 0.07067 0.0012902 0.0015521
beta4 0.945324 0.10718 0.0019568 0.0022698
beta5 0.078139 0.36039 0.0065799 0.0158843
beta6 0.951735 0.39612 0.0072321 0.0161983
kappa51 2.129596 0.22166 0.0040469 0.0106461
kappa61 -2.197417 0.24853 0.0045375 0.0122573
lambda11 0.734338 0.07409 0.0013527 0.0033106
lambda21 -0.736483 0.08285 0.0015127 0.0035505
lambda31 1.527181 0.14822 0.0027061 0.0069023
lambda41 -1.517229 0.16025 0.0029258 0.0066417
mu 0.062361 0.16611 0.0030328 0.0032710
sigma1 0.182383 0.03539 0.0006460 0.0008676
sigma2 0.309895 0.04363 0.0007966 0.0008775
sigma3 0.197108 0.09265 0.0016915 0.0026703
sigma4 0.548641 0.07684 0.0014029 0.0014900
sigma5 0.606646 0.51096 0.0093288 0.0346721
sigma6 1.058430 0.51137 0.0093363 0.0337731
2. Quantiles for each variable:
2.5% 25% 50% 75% 97.5%
beta2 4.88235 4.95842 4.996905 5.03529 5.1096
beta3 -0.13373 -0.04408 0.001406 0.05088 0.1418
beta4 0.74073 0.87268 0.946501 1.01743 1.1563
beta5 -0.68024 -0.14712 0.082675 0.30860 0.7820
beta6 0.19090 0.68793 0.942025 1.20350 1.7285
kappa51 1.73555 1.97474 2.114284 2.27036 2.5995
kappa61 -2.73185 -2.35638 -2.187416 -2.02858 -1.7572
lambda11 0.60816 0.68097 0.730254 0.78011 0.8980
lambda21 -0.91053 -0.78708 -0.730500 -0.67891 -0.5890
lambda31 1.26822 1.41957 1.518770 1.62056 1.8489
lambda41 -1.85994 -1.61766 -1.506186 -1.40227 -1.2386
mu -0.26345 -0.05091 0.059691 0.17004 0.3950
sigma1 0.10981 0.16147 0.183202 0.20442 0.2497
sigma2 0.23424 0.27936 0.306414 0.33644 0.4047
sigma3 0.03645 0.12488 0.198023 0.26107 0.3823
sigma4 0.42278 0.49283 0.540751 0.59475 0.7190
sigma5 0.03043 0.13175 0.430753 1.12118 1.5313
sigma6 0.04954 0.68329 1.246044 1.42383 1.7243

0に固定するのをbeta2としてもMCMCは収束するが、収束速度が落ちるような気がする。このあたり、データにあったモデリングが必要ということか。





コメント 0