多重共線性とか潜在変数とか共分散構造分析とか [統計]
多重共線性のことを考えることがあって、モデル選択では解決できなかった
が、潜在変数を導入してみれば、と、そうすると共分散構造分析(SEM)になるなぁ、とそういえば、
豊田(2008)
ではSEMも扱っていたな(豊田先生だし)、ということでちょっと実験してみた。
豊田ほか(1992)
の107ページにあるような関係を想定。
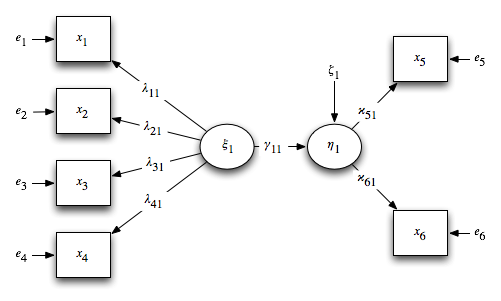
x1〜x4を胸高直径とか樹高とかに、x5〜x6を伸長成長量とか肥大成長量とかにして、ξ1をサイズ、&eta1を成長とかとイメージしてみることもできる。
データを用意してみる。
set.seed(123) N <- 40 x1 <- rnorm(N, 0, 1) x2 <- x1 + rnorm(N, 0, 0.3) x3 <- 2 * x1 + rnorm(N, 0, 0.5) x4 <- x3 + rnorm(N, 0, 0.5) x5 <- 4 * x1 + rnorm(N, 0, 0.8) x6 <- 8 * x1 + rnorm(N, 0, 1.2) data <- cbind(x1, x2, x3, x4, x5, x6) pairs(data)
まずは通常の共分散構造分析をやってみる。library(sem)を使用。
library(sem)
sem.model <- specify.model("model.txt")
sem.result <- sem(sem.model, cov(data), N)
summary(sem.result)
model.txt
x1 <- xi1, lambda11, NA x2 <- xi1, lambda21, NA x3 <- xi1, lambda31, NA x4 <- xi1, lambda41, NA x5 <- eta1, kappa51, NA x6 <- eta1, kappa61, NA eta1 <- xi1, NA, 1 x1 <-> x1, psi1, NA x2 <-> x2, psi2, NA x3 <-> x3, psi3, NA x4 <-> x4, psi4, NA x5 <-> x5, psi5, NA x6 <-> x6, psi6, NA eta1 <-> eta1, NA, 1 xi1 <-> xi1, NA, 1
結果
Model Chisquare = 118.76 Df = 9 Pr(>Chisq) = 0
Chisquare (null model) = 588.88 Df = 15
Goodness-of-fit index = 0.68907
Adjusted goodness-of-fit index = 0.27449
RMSEA index = 0.55921 90% CI: (NA, NA)
Bentler-Bonnett NFI = 0.79833
Tucker-Lewis NNFI = 0.68123
Bentler CFI = 0.80874
SRMR = 0.44166
BIC = 85.562
Normalized Residuals
Min. 1st Qu. Median Mean 3rd Qu. Max.
2.13 2.60 2.80 3.44 4.48 4.75
Parameter Estimates
Estimate Std Error z value Pr(>|z|)
lambda11 0.7007011 0.065754 10.65642 0.0000e+00 x1 <--- xi1
lambda21 0.7164996 0.075339 9.51036 0.0000e+00 x2 <--- xi1
lambda31 1.4131920 0.139424 10.13595 0.0000e+00 x3 <--- xi1
lambda41 1.4493645 0.153612 9.43523 0.0000e+00 x4 <--- xi1
kappa51 1.8737647 0.275043 6.81262 9.5837e-12 x5 <--- eta1
kappa61 4.1159250 0.447021 9.20745 0.0000e+00 x6 <--- eta1
psi1 0.0085807 0.011788 0.72791 4.6667e-01 x1 <--> x1
psi2 0.0821155 0.020632 3.98003 6.8906e-05 x2 <--> x2
psi3 0.1373468 0.060341 2.27618 2.2835e-02 x3 <--> x3
psi4 0.3225484 0.117951 2.73459 6.2459e-03 x4 <--> x4
psi5 2.0775278 2.321664 0.89484 3.7087e-01 x5 <--> x5
psi6 -3.8338943 6.884330 -0.55690 5.7759e-01 x6 <--> x6
Iterations = 77
psi6が、分散なのに負の値になっているのはサンプルサイズが小さいせいであろう。豊田(2008)の62ページに説明あり。GFIの値もいまひとつかふたつか、それ以上。
つづいてMCMC。
library(rjags)
mcmc.data <- list(N = N, x1 = x1, x2 = x2, x3 = x3,
x4 = x4, x5 = x5, x6 = x6)
init1 <- list(lambda11 = 1, lambda21 = 1,
lambda31 = 1, lambda41 = 1,
kappa51 = 1, kappa61 = 1,
tau1 = 1, tau2 = 1, tau3 = 1,
tau4 = 1, tau5 = 1, tau6 = 1,
m = 1)
init2 <- list(lambda11 = 5, lambda21 = 5,
lambda31 = 5, lambda41 = 5,
kappa51 = 5, kappa61 = 5,
tau1 = 2, tau2 = 2, tau3 = 2,
tau4 = 2, tau5 = 2, tau6 = 2,
m = 1)
init3 <- list(lambda11 = 10, lambda21 = 10,
lambda31 = 10, lambda41 = 10,
kappa51 = 10, kappa61 = 10,
tau1 = 0.1, tau2 = 0.1, tau3 = 0.1,
tau4 = 0.1, tau5 = 0.1, tau6 = 0.1,
m = 1)
mcmc.model <- jags.model("sem.bug", mcmc.data,
inits = list(init1, init2, init3),
nchain = 3,
n.adapt = 30000)
vars <- c("lambda11", "lambda21", "lambda31",
"lambda41", "kappa51", "kappa61",
"e1", "e2", "e3", "e4", "e5", "e6",
"sigma1", "sigma2", "sigma3", "sigma4",
"sigma5", "sigma6", "m")
mcmc.samples <- coda.samples(mcmc.model, vars,
n.iter = 20000, thin = 20)
sem.bug
var
N, # number of data
x1[N], x2[N], x3[N], x4[N], x5[N], x6[N], # data
xi1[N], eta1[N], # latent variables
lambda11, lambda21, lambda31, lambda41, # parameters
kappa51, kappa61, m,
tau1, tau2, tau3, tau4, tau5, tau6,
sigma1, sigma2, sigma3, sigma4, sigma5, sigma6
model {
for (i in 1:N) {
x1[i] ~ dnorm(mu1[i], tau1);
mu1[i] <- lambda11 * xi1[i] + e1;
x2[i] ~ dnorm(mu2[i], tau2);
mu2[i] <- lambda21 * xi1[i] + e2;
x3[i] ~ dnorm(mu3[i], tau3);
mu3[i] <- lambda31 * xi1[i] + e3;
x4[i] ~ dnorm(mu4[i], tau4);
mu4[i] <- lambda41 * xi1[i] + e4;
x5[i] ~ dnorm(mu5[i], tau5);
mu5[i] <- kappa51 * eta1[i] + e5;
x6[i] ~ dnorm(mu6[i], tau6);
mu6[i] <- kappa61 * eta1[i] + e6;
eta1[i] ~ dnorm(mu[i], 1);
mu[i] <- xi1[i];
xi1[i] ~ dnorm(m, 1);
}
lambda11 ~ dnorm(0, 1.0E-6);
lambda21 ~ dnorm(0, 1.0E-6);
lambda31 ~ dnorm(0, 1.0E-6);
lambda41 ~ dnorm(0, 1.0E-6);
kappa51 ~ dnorm(0, 1.0E-6);
kappa61 ~ dnorm(0, 1.0E-6);
e1 ~ dnorm(0, 1.0E-6);
e2 ~ dnorm(0, 1.0E-6);
e3 ~ dnorm(0, 1.0E-6);
e4 ~ dnorm(0, 1.0E-6);
e5 ~ dnorm(0, 1.0E-6);
e6 ~ dnorm(0, 1.0E-6);
m ~ dgamma(1.0E-3, 1.0E-3);
tau1 ~ dgamma(1.0E-3, 1.0E-3);
tau2 ~ dgamma(1.0E-3, 1.0E-3);
tau3 ~ dgamma(1.0E-3, 1.0E-3);
tau4 ~ dgamma(1.0E-3, 1.0E-3);
tau5 ~ dgamma(1.0E-3, 1.0E-3);
tau6 ~ dgamma(1.0E-3, 1.0E-3);
sigma1 <- 1/sqrt(tau1);
sigma2 <- 1/sqrt(tau2);
sigma3 <- 1/sqrt(tau3);
sigma4 <- 1/sqrt(tau4);
sigma5 <- 1/sqrt(tau5);
sigma6 <- 1/sqrt(tau6);
}
結果
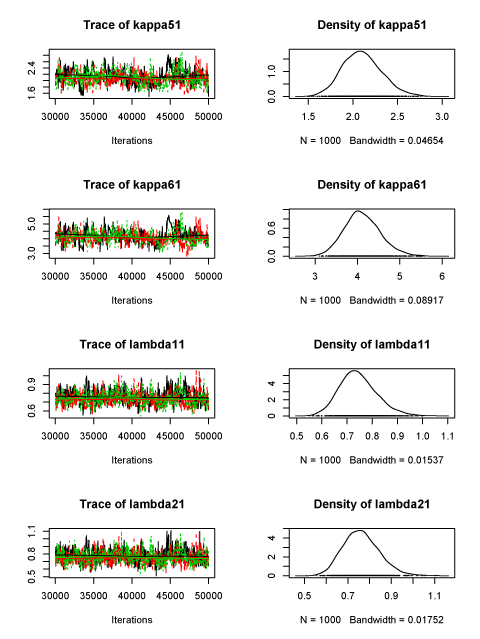
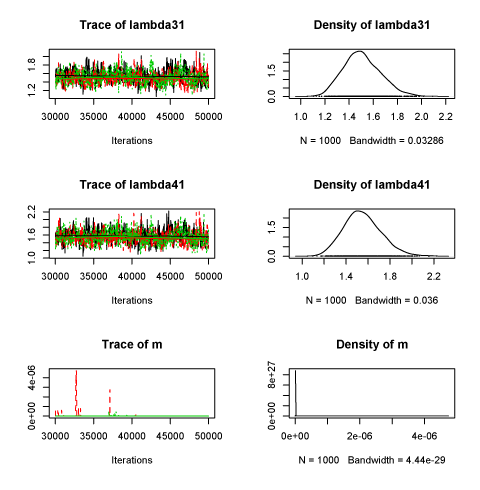
Iterations = 30020:50000
Thinning interval = 20
Number of chains = 3
Sample size per chain = 1000
1. Empirical mean and standard deviation for each variable,
plus standard error of the mean:
Mean SD Naive SE Time-series SE
e1 3.922e-02 1.194e-01 2.180e-03 7.337e-03
e2 3.809e-02 1.296e-01 2.366e-03 7.262e-03
e3 8.311e-02 2.470e-01 4.509e-03 1.485e-02
e4 3.357e-02 2.623e-01 4.788e-03 1.520e-02
e5 1.886e-01 4.902e-01 8.949e-03 3.381e-02
e6 3.599e-01 9.595e-01 1.752e-02 6.579e-02
kappa51 2.104e+00 2.177e-01 3.975e-03 1.351e-02
kappa61 4.115e+00 4.265e-01 7.787e-03 2.648e-02
lambda11 7.448e-01 7.451e-02 1.360e-03 3.682e-03
lambda21 7.618e-01 8.356e-02 1.526e-03 3.556e-03
lambda31 1.508e+00 1.551e-01 2.831e-03 7.326e-03
lambda41 1.550e+00 1.715e-01 3.131e-03 7.730e-03
m 1.296e-08 1.877e-07 3.426e-09 1.138e-08
sigma1 1.129e-01 4.183e-02 7.637e-04 1.051e-03
sigma2 3.035e-01 4.119e-02 7.520e-04 7.201e-04
sigma3 3.574e-01 7.041e-02 1.285e-03 1.627e-03
sigma4 5.663e-01 8.535e-02 1.558e-03 1.886e-03
sigma5 6.397e-01 3.731e-01 6.813e-03 2.701e-02
sigma6 1.036e+00 7.580e-01 1.384e-02 5.476e-02


パラメータが正負反転したりするところを、初期値でなんとかしたり、潜在変数に制約をつけるあたりで苦労した。mがほとんど0だが、0に固定してしまうと他の収束がわるくなったりで、モデルが要再考なのかも。





コメント 0